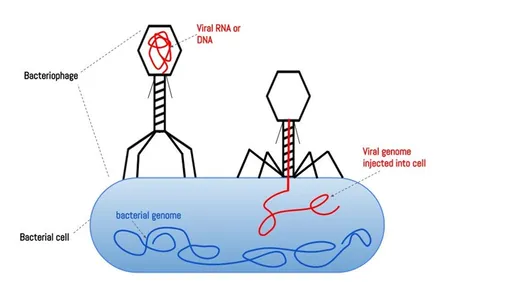
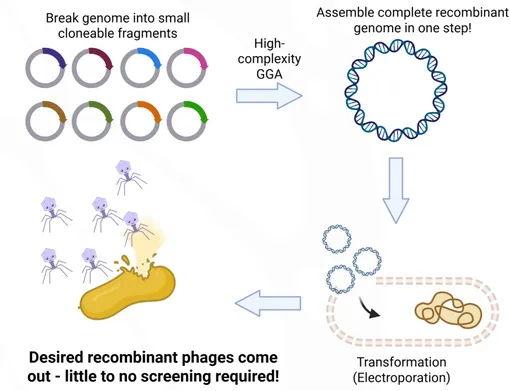
Традиционная терапия бактериофагами — вирусами, убивающими бактерии, — долгое время опиралась на поиск природных штаммов, что ограничивало возможности медицины в условиях глобального роста устойчивости к антибиотикам. Новая технология, опубликованная в журнале Proceedings of the National Academy of Sciences, позволяет собирать вирусный геном из десятков синтетических фрагментов, как из деталей Лего.
Ученые синтезировали искусственный вирус для борьбы с супербактериями
В ходе эксперимента ученые воссоздали фаг Pseudomonas aeruginosa из 28 фрагментов ДНК, попутно наделив его новыми свойствами. Путем точечных мутаций и вставок исследователи изменили структуру «хвостовых нитей» вируса, что позволило расширить спектр поражаемых им бактерий, а внедрение флуоресцентных маркеров дало возможность наблюдать за процессом инфекции в режиме реального времени.
Соавтор работы Энди Сиккема отмечает: «Даже в лучших случаях инженерная биология бактериофагов была чрезвычайно трудоемкой. Исследователи тратили целые карьеры на разработку процессов модификации конкретных моделей. Этот синтетический метод предлагает технологический скачок в простоте, безопасности и скорости, прокладывая путь к новым терапевтическим открытиям».
Оружие направленного действия
Главное преимущество платформы HC-GGA заключается в возможности редактировать вирус вне живой клетки. Ученым больше не нужно полагаться на трудоемкий скрининг или многократные итерации правок внутри патогенных бактерий.
Вместо этого они создают идеальный чертеж генома в цифровом виде и воплощают его в биохимической смеси. Этот подход снимает проблему токсичности длинных фрагментов ДНК, что раньше было серьезным препятствием.
Как образно выразился соавтор исследования Грег Ломан: «Моя лаборатория создает "необычные молотки", а затем ищет подходящие гвозди. В данном случае сообщество разработчиков фаговой терапии сказало нам: "Это именно тот молоток, которого мы ждали"». Теперь создание вирусов-по-требованию для лечения конкретного пациента становится вопросом недель, а не десятилетий.